Дегрон
Дегро́н (англ. Degron) — часть молекулы белка, которая регулирует скорость его разрушения (протеолиза). Известные на данный момент дегроны представляют собой короткие аминокислотные последовательности[1], структурные мотивы[2] или экспонированные из белковой глобулы аминокислотные остатки (часто остатки лизина[3] или аргинина[4]), располагающиеся в любом участке аминокислотной цепочки. Некоторые белки содержат несколько дегронов[2][5]. Дегроны выявлены у белков разнообразных организмов, начиная от N-концевых дегронов дрожжей[6] и кончая последовательностью PEST в орнитиндекарбоксилазе мыши[7]. Дегроны также были выявлены в белках прокариот[8].
Известные дегроны подразделяют на несколько групп, но даже дегроны одной группы очень вариабельны, хотя все они так или иначе влияют на деградацию белка[9][10][11]. Для работы некоторых дегронов (убиквитин-зависимых) необходимо, чтобы белок был мечен убиквитином, другие (убиквитин-независимые) работают независимо от мечения белка убиквитином[12][10][13].
Типы
Для функционирования убиквитин-зависимых дегронов необходимо полиубиквитинирование белка, которое направляет его на разрушение в протеасомы[14][15]. В некоторых случаях дегрон сам по себе служит сайтом полиубиквитинирования, как в случая белка β-катенина[16]. Так как детальный механизм участия дегрона в полиубиквитинировании известен далеко не всегда, дегроны признаются убиквитин-зависимыми, если при их удалении из белка уровень его убиквитинирования снижается, а при добавлении к белку уровень убиквитинирования, напротив, повышается[17][18].
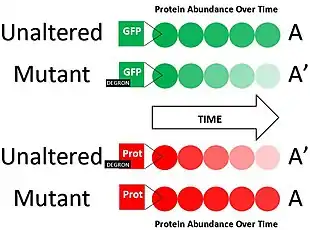
Вверху: исходная форма белка остаётся стабильнее формы, к которой пришит предполагаемый дегрон, и разрушается медленнее.
Внизу: форма белка с удалённым предполагаемым дегроном разрушается медленнее исходной формы
Функционирование убиквитин-независимых дегронов, напротив, не сопровождается полиубиквитинированием содержащих их белков. Например, дегрон IκBα (белка, участвующего в работе иммунной системы) не задействован в убиквитинировании, поскольку сшивка этого дегрона с зелёным флуоресцентным белком (GFP) не повышало уровень убиквитинирования последнего[2]. Однако детальный механизм того, как именно дегрон задействован в разрушении белка, в большинстве случаев неизвестен[19].
Идентификация
Существует три подхода, позволяющие идентифицировать дегрон в белке[2][18][19]. В первом методе последовательность, являющаяся вероятным дегроном, пришивается к стабильному белку (например, GFP), и далее сравнивается стабильность исходного белка и белка с пришитым вероятным дегроном[20]. Если пришитая последовательность действительно является дегроном, число молекул белка, к которому она пришита, будет со временем снижаться гораздо быстрее, чем в случае исходного белка[9][10][11]. Кроме того, из белка можно удалить последовательность, которая, вероятно, является дегроном, и сравнить стабильность белка с делецией с исходным белком. Если удалённая последовательность является дегроном, белковые молекулы, лишённые её, окажутся стабильнее исходного белка[9][10][11].
Третий подход используется для установления зависимости дегрона от убиквитинирования, когда с помощью описанных выше приёмов дегронная природа последовательности уже была подтверждена. В этом подходе сравнивается степень убиквитинирования исходного белка и мутантной формы, лишённой дегрона[2][7][19].
Примечания
- Cho S., Dreyfuss G. A degron created by SMN2 exon 7 skipping is a principal contributor to spinal muscular atrophy severity. (англ.) // Genes & Development. — 2010. — 1 March (vol. 24, no. 5). — P. 438—442. — doi:10.1101/gad.1884910. — PMID 20194437.
- Fortmann K. T., Lewis R. D., Ngo K. A., Fagerlund R., Hoffmann A. A Regulated, Ubiquitin-Independent Degron in IκBα. (англ.) // Journal Of Molecular Biology. — 2015. — 28 August (vol. 427, no. 17). — P. 2748—2756. — doi:10.1016/j.jmb.2015.07.008. — PMID 26191773.
- Dohmen R. J., Wu P., Varshavsky A. Heat-inducible degron: a method for constructing temperature-sensitive mutants. (англ.) // Science (New York, N.Y.). — 1994. — 4 March (vol. 263, no. 5151). — P. 1273—1276. — PMID 8122109.
- Varshavsky A. The N-end rule: functions, mysteries, uses. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1996. — 29 October (vol. 93, no. 22). — P. 12142—12149. — PMID 8901547.
- Kanarek N., London N., Schueler-Furman O., Ben-Neriah Y. Ubiquitination and degradation of the inhibitors of NF-kappaB. (англ.) // Cold Spring Harbor Perspectives In Biology. — 2010. — February (vol. 2, no. 2). — P. 000166—000166. — doi:10.1101/cshperspect.a000166. — PMID 20182612.
- Bachmair A., Finley D., Varshavsky A. In vivo half-life of a protein is a function of its amino-terminal residue. (англ.) // Science (New York, N.Y.). — 1986. — 10 October (vol. 234, no. 4773). — P. 179—186. — PMID 3018930.
- Loetscher P., Pratt G., Rechsteiner M. The C terminus of mouse ornithine decarboxylase confers rapid degradation on dihydrofolate reductase. Support for the pest hypothesis. (англ.) // The Journal Of Biological Chemistry. — 1991. — 15 June (vol. 266, no. 17). — P. 11213—11220. — PMID 2040628.
- Burns K. E., Liu W. T., Boshoff H. I., Dorrestein P. C., Barry CE 3rd. Proteasomal protein degradation in Mycobacteria is dependent upon a prokaryotic ubiquitin-like protein. (англ.) // The Journal Of Biological Chemistry. — 2009. — 30 January (vol. 284, no. 5). — P. 3069—3075. — doi:10.1074/jbc.M808032200. — PMID 19028679.
- Ravid T., Hochstrasser M. Diversity of degradation signals in the ubiquitin-proteasome system. (англ.) // Nature Reviews. Molecular Cell Biology. — 2008. — September (vol. 9, no. 9). — P. 679—690. — doi:10.1038/nrm2468. — PMID 18698327.
- Erales J., Coffino P. Ubiquitin-independent proteasomal degradation. (англ.) // Biochimica Et Biophysica Acta. — 2014. — January (vol. 1843, no. 1). — P. 216—221. — doi:10.1016/j.bbamcr.2013.05.008. — PMID 23684952.
- Jariel-Encontre I., Bossis G., Piechaczyk M. Ubiquitin-independent degradation of proteins by the proteasome. (англ.) // Biochimica Et Biophysica Acta. — 2008. — December (vol. 1786, no. 2). — P. 153—177. — doi:10.1016/j.bbcan.2008.05.004. — PMID 18558098.
- Asher G., Tsvetkov P., Kahana C., Shaul Y. A mechanism of ubiquitin-independent proteasomal degradation of the tumor suppressors p53 and p73. (англ.) // Genes & Development. — 2005. — 1 February (vol. 19, no. 3). — P. 316—321. — doi:10.1101/gad.319905. — PMID 15687255.
- Hochstrasser M. Ubiquitin-dependent protein degradation. (англ.) // Annual Review Of Genetics. — 1996. — Vol. 30. — P. 405—439. — doi:10.1146/annurev.genet.30.1.405. — PMID 8982460.
- Coux O., Tanaka K., Goldberg A. L. Structure and functions of the 20S and 26S proteasomes. (англ.) // Annual Review Of Biochemistry. — 1996. — Vol. 65. — P. 801—847. — doi:10.1146/annurev.bi.65.070196.004101. — PMID 8811196.
- Lecker S. H., Goldberg A. L., Mitch W. E. Protein degradation by the ubiquitin-proteasome pathway in normal and disease states. (англ.) // Journal Of The American Society Of Nephrology : JASN. — 2006. — July (vol. 17, no. 7). — P. 1807—1819. — doi:10.1681/ASN.2006010083. — PMID 16738015.
- Melvin A. T., Woss G. S., Park J. H., Dumberger L. D., Waters M. L., Allbritton N. L. A comparative analysis of the ubiquitination kinetics of multiple degrons to identify an ideal targeting sequence for a proteasome reporter. (англ.) // PloS One. — 2013. — Vol. 8, no. 10. — P. e78082—78082. — doi:10.1371/journal.pone.0078082. — PMID 24205101.
- Wang Y., Guan S., Acharya P., Koop D. R., Liu Y., Liao M., Burlingame A. L., Correia M. A. Ubiquitin-dependent proteasomal degradation of human liver cytochrome P450 2E1: identification of sites targeted for phosphorylation and ubiquitination. (англ.) // The Journal Of Biological Chemistry. — 2011. — 18 March (vol. 286, no. 11). — P. 9443—9456. — doi:10.1074/jbc.M110.176685. — PMID 21209460.
- Ju D., Xie Y. Identification of the preferential ubiquitination site and ubiquitin-dependent degradation signal of Rpn4. (англ.) // The Journal Of Biological Chemistry. — 2006. — 21 April (vol. 281, no. 16). — P. 10657—10662. — doi:10.1074/jbc.M513790200. — PMID 16492666.
- Schrader E. K., Harstad K. G., Matouschek A. Targeting proteins for degradation. (англ.) // Nature Chemical Biology. — 2009. — November (vol. 5, no. 11). — P. 815—822. — doi:10.1038/nchembio.250. — PMID 19841631.
- Li X., Zhao X., Fang Y., Jiang X., Duong T., Fan C., Huang C. C., Kain S. R. Generation of destabilized green fluorescent protein as a transcription reporter. (англ.) // The Journal Of Biological Chemistry. — 1998. — 25 December (vol. 273, no. 52). — P. 34970—34975. — PMID 9857028.